Capítulo 10. El flujo de información genética: los caminos del DNA a la proteína
La evolución del concepto de gen
1. A partir de la década de 1940, el concepto de gen evolucionó desde la formulación "un gen: una enzima" a "un gen: una cadena polipeptídica". Sin embargo, ninguna de estas definiciones se ajusta por completo al concepto actual de gen.
El flujo de información dentro de la célula
2. El "dogma central de la biología", definido en 1957 por Francis Crick, establece que la información genética fluye en el siguiente sentido: DNA → RNA→ proteínas. Esto es verdad en la mayoría de los casos; sin embargo, el material genético de algunos virus está formado por RNA que luego es usado como molde para producir DNA.
El código genético
3. El código genético consiste en la asignación de tripletes de nucleótidos (codones) en el RNA mensajero (mRNA) a cada uno de los aminoácidos que formarán una cadena polipeptídica.
4. Existen 64 combinaciones posibles de codones. El código es redundante, porque los 20 aminoácidos usualmente presentes en los seres vivos son codificados por 61 de estas combinaciones. Los tres codones restantes actúan como señales de terminación de la traducción.
5. Con muy pocas excepciones, el código genético es el mismo en casi todos los seres vivos.
Fig. 10-4. El código genético
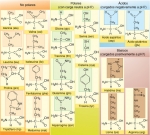

(a) Los 20 aminoácidos que comúnmente constituyen las proteínas. (b) El código genético consiste en 64 combinaciones de tripletes (codones) y sus aminoácidos correspondientes. Los codones que se muestran aquí son los que puede presentar la molécula de mRNA. De los 64 codones, 61 especifican aminoácidos particulares. Los otros 3 codones son señales de detención, que determinan la finalización de la cadena polipeptídica. Dado que los 61 tripletes codifican para 20 aminoácidos, hay codones "sinónimos" como, por ejemplo, los 6 tripletes diferentes que incorporan leucina (la mayoría de los sinónimos, como se puede ver, difieren sólo en el tercer nucleótido). Sin embargo, la afirmación inversa no es válida: cada codón especifica solamente un aminoácido.
La transcripción: del DNA al RNA
6. La transcripción es el proceso de síntesis de RNA a partir de DNA. Sigue el mismo principio de apareamiento de bases que la replicación del DNA, pero se reemplaza la timina por el uracilo. En cada transcripción, sólo una de las cadenas del DNA se transcribe. La RNA polimerasa cataliza la adición de ribonucleótidos al extremo 3´ de la cadena de RNA, de modo que esta última es antiparalela a la cadena molde de DNA.
Fig. 10-6. Transcripción del DNA
![]()
En la región del promotor, punto de unión de la enzima RNA polimerasa, la doble hélice de DNA se abre y, a medida que la RNA polimerasa avanza a lo largo de la molécula de DNA, se separan las dos cadenas. Los ribonucleótidos, que constituyen los bloques estructurales, se ensamblan en la dirección 5' a 3' a medida que la enzima lee la cadena molde de DNA. Nótese que la cadena de RNA recién sintetizada es complementaria, no idéntica, a la cadena molde a partir de la cual se transcribe; su secuencia, sin embargo, es idéntica a la cadena codificante de DNA (no transcrita), excepto por un detalle: en el RNA, la timina (T) se reemplaza por uracilo (U). El RNA recién sintetizado se separa de la cadena molde de DNA.
7. La RNA polimerasa no necesita un cebador para iniciar la síntesis. Se une al DNA en una secuencia específica, el promotor, que define el punto de inicio de la transcripción y su dirección.
8. En los procariontes, el proceso de transcripción continúa hasta que la polimerasa encuentra una secuencia que constituye la señal de terminación. En los eucariontes, el proceso termina cuando el RNA es cortado en una secuencia específica. Al finalizar la transcripción, la RNA polimerasa se detiene y libera la cadena molde de DNA y el mRNA sintetizado.
9. En los eucariontes, los transcritos primarios sufren diversas modificaciones durante la transcripción. Entre ellas se encuentran la adición del CAP, la poliadenilación y el splicing. Este último proceso consiste en el corte y la eliminación de ciertas secuencias, los intrones, y el posterior empalme de las secuencias restantes, los exones. Sólo los exones forman parte del mRNA maduro. Un mismo transcrito primario puede ser procesado por splicing de distintas maneras. Este empalme alternativo permite que una molécula de mRNA inmadura pueda originar diferentes moléculas de mRNA maduro.
Fig. 10-8. Procesamiento del mRNA en eucariontes
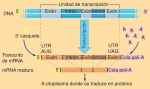
La información genética codificada en el DNA se transcribe a una copia de RNA (transcripto primario). Esta copia se modifica en forma cotranscripcional con la adición del casquete 5' (CAP), el corte de los intrones y el empalme de los exones (splicing) y, finalmente con la adición de la cola de poliA. A ambos extremos del mensajero hay secuencias no traducibles, denominadas extremos 5´UTR (región no traducible que abarca desde el CAP hasta el codón de iniciación) y extremos 3´UTR (región no traducible que abarca desde el codón de terminación hasta la cola de PoliA). En esta figura, el splicing se produce luego de la adición de la cola de poli-A, sin embargo, muchas veces el proceso de corte y empalme ocurre antes de que haya concluido la transcripción. El mRNA maduro luego se dirige al citoplasma, donde se traduce a proteínas.
10. En el ciliado de agua dulce Tetrahymena, el intrón inmaduro actúa como catalizador de la escisión, produciendo un empalme autocatalítico. A este RNA con función de enzima se lo llama ribozima.
La traducción: del RNA al polipéptido
11. La traducción es la conversión de la secuencia de nucleótidos del RNA en la secuencia de aminoácidos de un polipéptido. En este proceso participan los mRNA, los RNA ribosómicos (rRNA) y los RNA de transferencia (tRNA).
12. Los ribosomas están formados por rRNA y proteínas. Cada uno está formado por dos subunidades de diferente tamaño que, además, en los procariontes son más pequeñas que en los eucariontes.
13. El mRNA y el tRNA iniciador se unen a la subunidad ribosómica menor. Luego se les une la subunidad mayor y cataliza la unión peptídica entre aminoácidos. En la subunidad mayor existen tres sitios a los que se une el tRNA: el sitio A (aminoacílico), el sitio P (peptidílico) y el sitio E (de salida).
14. Los tRNA son moléculas pequeñas, con una estructura secundaria semejante a la hoja de un trébol, que presentan dos sitios de unión. Uno de ellos es el anticodón, que se aparea con el codón del mRNA. El otro sitio, ubicado en el extremo 3´, se acopla a un aminoácido particular en forma muy específica. Así, los tRNA permiten la alineación de los aminoácidos de acuerdo con la secuencia de nucleótidos del mRNA.
15. El grupo de enzimas aminoacil-tRNA sintetasas catalizan la unión entre el aminoácido y el tRNA y forman el complejo aminoacil-tRNA. Este complejo se une a la molécula de mRNA, apareando el anticodón con el codón del mRNA en forma antiparalela. Así, el tRNA coloca al aminoácido específico en su lugar. El enlace entre el aminoácido y el tRNA se rompe cuando se forma el enlace entre el aminoácido recién llegado y el último de la cadena polipeptídica en crecimiento.
16. En los procariontes, el proceso de traducción comienza antes de que haya finalizado el de transcripción. En los eucariontes, ambos procesos están separados en el tiempo y en el espacio: la transcripción ocurre en el núcleo y la traducción, en el citoplasma.
17. Tanto en procariontes como en eucariontes, la síntesis de polipéptidos ocurre en tres etapas: iniciación, elongación y terminación.
18. Hacia el final del mRNA hay un codón que actúa como señal de terminación. No existe ningún tRNA que tenga un anticodón que se aparee con este codón. Existen, en cambio, factores de liberación que se unen al codón de terminación y provocan la separación del polipéptido y el tRNA. Finalmente, las dos subunidades ribosómicas también se separan.
Fig. 10-13. Síntesis de un polipéptido en procariontes
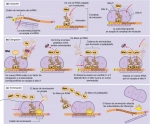
(a) Iniciación. La subunidad ribosómica más pequeña (menor) se une al extremo 5' de una molécula de mRNA. La primera molécula de tRNA, que lleva el aminoácido modificado fMet, se acopla con el codón iniciador AUG de la molécula de mRNA. La subunidad ribosómica más grande (mayor) se ubica en su lugar, el complejo tRNA-fMet ocupa el sitio P (peptidílico). El complejo de iniciación ahora está completo. (b) Elongación. Un segundo tRNA, cargando su aminoácido correspondiente, valina en este caso, se coloca en el sitio A y su anticodón se acopla con el mRNA. Para que el aminoacil-tRNA ingrese en el sitio A debe unirse antes a una proteína llamada factor de elongación, que en su forma activa está unida al GTP. Al aparearse el tRNA con el mRNA, se dispara la hidrólisis del GTP por parte del factor de elongación, que luego se disocia, lo cual permite que el aminoacil-tRNA permanezca unido por un corto período al mRNA. A continuación se forma un enlace peptídico entre los dos aminoácidos reunidos en el ribosoma. Al mismo tiempo, el enlace entre el primer aminoácido y su tRNA se rompe. El ribosoma se mueve entonces a lo largo de la cadena de mRNA en dirección 5' a 3'. El segundo tRNA, con el dipéptido unido, se mueve desde el sitio A al sitio P y el primer tRNA pasa al sitio E y luego se desprende del ribosoma. Un tercer aminoacil-tRNA, que en este caso porta el aminoácido fenilalanina, se coloca en el sitio A y se forma otro enlace peptídico. La cadena peptídica naciente siempre está unida al tRNA que se está moviendo del sitio A al sitio P y el tRNA entrante que lleva el siguiente aminoácido siempre ocupa el sitio A. Este paso se repite una y otra vez hasta que se completa el polipéptido. (c) Terminación. Cuando el ribosoma alcanza un codón de terminación (en este ejemplo, UGA), el sitio A es ocupado por factores de liberación que hacen que la cadena polipeptídica se escinda del último tRNA y que las dos subunidades del ribosoma se disocien.
19. Las proteínas "chaperonas" ayudan a las cadenas polipeptídicas a plegarse. Finalizado este proceso, las nuevas proteínas viajan al medio extracelular o a los distintos compartimientos celulares, según el tipo de señales que posean.
Una redefinición de las mutaciones
20. Una mutación es un cambio en la secuencia o en el número de nucleótidos en el DNA de una célula. Sólo las mutaciones que ocurren en los gametos se transmiten a la descendencia. Las mutaciones puntuales implican la sustitución de un nucleótido por otro. La adición o la sustracción (deleción) de nucleótidos provoca el corrimiento del marco de lectura y, por consiguiente, la aparición de una proteína nueva que casi siempre resulta defectuosa.
Una revisión del concepto de gen
21. Actualmente se considera que un gen es un segmento de DNA que se encuentra a continuación de un promotor y que puede ser transcrito por una RNA polimerasa, originando un RNA funcional.
