Capítulo 9. Las bases químicas de la herencia: el DNA y su replicación
La química de la herencia
1. El DNA fue aislado por primera vez en 1869 por Friedrich Miescher. Luego se aislaron los distintos tipos de bases nitrogenadas que conforman el DNA y se demostró que el cromosoma eucarionte contenía DNA y proteínas en cantidades aproximadamente iguales. La complejidad de las proteínas llevó a pensar que ellas eran los genes.
La pista del DNA
2. El DNA es una molécula con forma de hélice que contiene la información genética de los seres vivos. Está formado por cuatro nucleótidos, compuestos por una base nitrogenada, un azúcar de desoxirribosa y un grupo fosfato. Los nucleótidos están constituidos por dos clases de bases nitrogenadas: las purinas adenina (A) y guanina (G), y las pirimidinas citosina (C) y timina (T). Entre los individuos de una misma especie, el DNA tiene igual proporción de A que de T, e igual proporción de G que de C. Todo esto se sabía a comienzos de la década de 1950.
El modelo de Watson y Crick
3. En 1953, usando la información disponible, James Watson y Francis Crick construyeron el modelo de estructura del DNA que se considera el más explicativo en la actualidad.
4. En el modelo de Watson y Crick, la molécula de DNA es una doble hélice formada por dos cadenas de nucleótidos apareadas. En cada cadena, los nucleótidos se pueden acoplar en cualquier orden o secuencia. Además, las adeninas de una cadena sólo se pueden aparear con timinas de la otra, las guaninas sólo con citosinas, y viceversa en ambos casos. El apareamiento se mantiene estable mediante puentes de hidrógeno entre los nucleótidos enfrentados. Las cadenas tienen dirección, pues los grupos fosfato forman un puente entre el quinto carbono del azúcar de un nucleótido y el tercer carbono del azúcar del siguiente, que determina un extremo 3' y otro 5'. Las dos cadenas apareadas corren en direcciones opuestas (son antiparalelas).
Fig. 9-7. La doble hélice del DNA
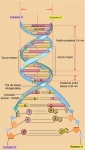
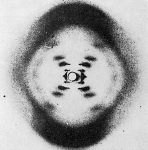
(a) El armazón de la hélice está compuesto por las unidades azúcar-fosfato de los nucleótidos. Los peldaños están formados por bases nitrogenadas, las purinas adenina y guanina -con una estructura de anillo doble- y las pirimidinas timina y citosina más pequeñas, con su estructura de anillo simple. Cada peldaño está formado por dos bases, una purina enfrentada a una pirimidina. Dos purinas combinadas tendrían más de 2 nanómetros y dos pirimidinas no alcanzarían para cubrir esta distancia. Pero una purina apareada en cada peldaño con una pirimidina mantienen un diámetro constante de 2 nanómetros en toda la longitud de la molécula. (b) Fotografía de difracción de rayos X del DNA, tomada por Rosalind Franklin, que resultó decisiva para la dilucidación de la estructura del DNA. Las reflexiones que se cruzan en el medio indican que la molécula es una hélice. Las regiones muy oscuras en las partes superior e inferior se deben a las bases, estrechamente apiladas, perpendiculares al eje de la hélice. El conocimiento de las distancias entre los átomos, determinadas con fotografías de difracción de rayos X como ésta, fue crucial para establecer la estructura de la molécula de DNA.
El mecanismo de replicación del DNA
5. La replicación del DNA es semiconservativa: la doble hélice se abre y cada cadena sirve de molde para la síntesis de una nueva cadena. Así se producen dos réplicas exactas de la molécula original.
6. La replicación comienza en una secuencia específica de nucleótidos llamada origen de la replicación. Los cromosomas procariontes tienen un solo origen de replicación; los eucariontes, varios.
7. En la replicación intervienen proteínas iniciadoras y enzimas que separan las dos cadenas de DNA. Las cadenas nuevas son sintetizadas por la DNA polimerasa III, que para comenzar su actividad requiere la presencia de un cebador (segmento de RNA sobre el cual inicia la síntesis). Al separarse las dos cadenas originales, se forman dos estructuras en forma de Y, llamadas horquillas de replicación. La replicación avanza en forma bidireccional, porque la síntesis y las dos horquillas de replicación se producen en direcciones opuestas desde un único origen.
8. La cadena 5' a 3' se sintetiza en forma continua como una sola unidad y se denomina adelantada; la cadena 3' a 5' se sintetiza de manera discontinua, como una serie de fragmentos llamados de Okazaki y se llama cadena retrasada. Cada fragmento de Okazaki es sintetizado en la dirección 5' a 3' y requiere un cebador. Luego, el RNA del cebador es reemplazado por DNA y la enzima ligasa une todos los fragmentos.
Fig. 9-13. Mecanismo de replicación del DNA
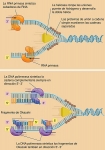
Las dos cadenas de la doble hélice de DNA se separan y sirven como moldes para la síntesis de nuevas cadenas complementarias. Las helicasas, enzimas que operan en las horquillas de replicación, separan las dos cadenas de la doble hélice original. Las proteínas de unión a cadena simple estabilizan las cadenas abiertas. La DNA polimerasa III cataliza la adición de nucleótidos a ambas cadenas operando sólo en dirección 5' a 3'. Para comenzar a añadir nucleótidos, esta enzima requiere la presencia de un cebador de RNA, unido por puentes de hidrógeno a la cadena molde que luego es reemplazado por nucleótidos de DNA. El cebador de RNA es sintetizado por la RNA primasa. La cadena adelantada se sintetiza en la dirección 5' a 3' en forma continua. En este caso, el único cebador de RNA está situado en el origen de replicación, que no es visible en este esquema. La cadena rezagada también se sintetiza en la dirección 5' a 3', a pesar de que esta dirección es opuesta a la del movimiento de la horquilla de replicación. El problema se resuelve mediante la síntesis discontinua de una serie de fragmentos, los fragmentos de Okazaki. Cuando un fragmento de Okazaki ha crecido lo suficiente como para encontrar un cebador de RNA por delante de él, la DNA polimerasa I reemplaza a los nucleótidos de RNA del cebador con nucleótidos de DNA. Luego, la DNA ligasa conecta cada fragmento con el fragmento contiguo recién sintetizado en la cadena.
9. El telómero es una secuencia fija y repetida de nucleótidos, presente en los extremos de los cromosomas lineales de las células eucariontes. En cada ciclo de replicación los telómeros se acortan, porque la DNA polimerasa no puede sintetizar DNA desde un extremo abierto. La enzima telomerasa compensa la pérdida prolongando los extremos.
Fig. 9-14. Alargamiento de los extremos de los cromosomas eucariontes por la enzima telomerasa
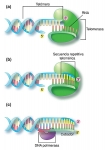
(a) El fragmento de RNA que porta la telomerasa se aparea con los últimos nucleótidos del telómero. (b) La porción proteica de la enzima cataliza la síntesis del DNA complementario al molde de RNA, elongando de este modo el extremo del cromosoma. (c) La RNA primasa y la DNA polimerasa sintetizan la cadena complementaria a la sintetizada por la telomerasa. El cebador sintetizado por la primasa luego es eliminado.
10. Muchos errores espontáneos que se producen durante la síntesis del DNA son reparados de inmediato por la propia DNA polimerasa III. Otros mecanismos de corrección realizan controles permanentes y reparan daños en el DNA. Las mutaciones son errores que quedan sin reparar y se transmiten a las células hijas.
11. La energía necesaria para que se produzcan las reacciones catalizadas por la DNA polimerasa es aportada por los enlaces de los fosfatos que forman parte de los nucleótidos.
La DNA polimerasa como herramienta de multiplicación: PCR
12. La reacción en cadena de la polimerasa permite multiplicar enormemente el número de moléculas de DNA de una muestra desconocida de tamaño ínfimo. Este método consiste en una serie de ciclos de calentamiento y enfriado que producen la desnaturalización, el copiado y el reapareamiento in vitro de la molécula de DNA. La síntesis de DNA es realizada por la Taq DNA polimerasa, una enzima de origen bacteriano que tolera la temperatura de desnaturalización del DNA. Esta técnica se utiliza para la detección de enfermedades hereditarias, identificación de personas, clonado de genes y pruebas de paternidad.
Fig. 9-15. La reacción en cadena de la polimerasa (PCR)

(a) La mezcla de reacción contiene la secuencia de DNA que se quiere amplificar, dos oligonucleótidos sintéticos (P1 y P2) que servirán como cebadores, una DNA polimerasa termoestable (llamada Taq DNA polimerasa) y los cuatro desoxirribonucleótidos dATP, dGTP, dCTP y dTTP. (b) Durante la desnaturalización, que se realiza por calentamiento de la mezcla a 95 ºC, se separan las dos cadenas del DNA molde. (c) La temperatura de incubación se reduce para permitir el apareamiento de las bases de ambos cebadores en el sitio donde encuentran una secuencia complementaria. (d) La mezcla se calienta a 72 ºC, temperatura a la cual la DNA polimerasa (Taq) extiende la cadena complementaria a partir del extremo 3' de los cebadores. Al finalizar cada ciclo (e), la cantidad de DNA molde disponible para el ciclo siguiente (f, g, y h) se duplica.
El DNA como portador de información
13. En el DNA, la información se encuentra en el ordenamiento lineal o secuencia de las cuatro bases que lo componen. La estructura del DNA puede dar cuenta de la enorme diversidad de los seres vivos
